広島大学大学院統合生命科学研究科
数理生命科学プログラム データ駆動生物学研究室
教授 本田直樹
E-mail:nhonda*hiroshima-u.ac.jp
Tel:082-424-7336
(注: *は半角@に置き換えてください)
本研究成果のポイント
- 1細胞計測データから遺伝子発現の空間パターンを再構成する機械学習(Perler)を開発
- 従来法と比べて、高精度かつ頑健な遺伝子発現の空間パターンの再構成に成功
- 今後の発生生物学などの基礎研究ならびに再生医療などへの応用に期待
概要
広島大学大学院統合生命科学研究科データ駆動生物学研究室の本田直樹教授(兼任:京都大学生命科学研究科特命教授、生命創成探究センター客員教授)、京都大学の大河内康之さん(医学部生・MD研究者育成プログラム)、坂口峻太さん(生命科学研究科大学院生)、中江健特定助教(情報学研究科)、近藤武史特定助教(生命科学研究科)らからなる共同研究グループは、1細胞RNAシーケンシング(RNA-seq)法で計測された遺伝子発現データから、遺伝子発現の空間的パターンをあたかもパズルを解くかのように再構成する機械学習法(Perler)を開発しました。
この提案手法は、従来法と比べて高精度かつ頑健に空間的遺伝子発現パターンを再構成することが示されました。正確な空間遺伝子発現パターンの再構成が実現されたことにより、発生過程における形作りや多細胞からなる組織機能の理解にも貢献することが期待されます。さらに、再生医療として作成されたミニ臓器が、実際の体内臓器をどの程度再現しているのかなどの評価に応用されることが期待されます。
本研究の成果は、Nature Communications誌(電子版)に掲載されました。
発表内容
【背景】
近年の生物学では、組織中の個々の細胞における全ての遺伝子の発現量を網羅的に調べることができる計測技術である1細胞RNA-seq法が盛んに利用されています。しかしながら、1細胞RNA-seq法では組織を1細胞までバラバラにしてしまうため、それぞれの細胞の組織中での位置情報が失われてしまうという欠点がありました。つまり、1細胞RNA-seq法を利用すると、組織における遺伝子発現の多様性を調べることはできても、空間における細胞間の相互関係を知ることはできないという問題がありました。この問題を解決するために、少数の遺伝子に対して空間的遺伝子発現パターンを調べることができる in situ hybridization(ISH)法のデータを参照することで、計算論的に網羅的な空間的遺伝子発現パターンを再構成するアプローチが注目されています(図1)。これまでこのようなアプローチを利用して、様々な組織(ショウジョウバエ胚やゼブラフィッシュ胚、マウス肝臓、マウス大脳皮質)における空間的遺伝子発現パターンが再構成されてきましたが、従来法の再構成精度は満足なものではありませんでした。
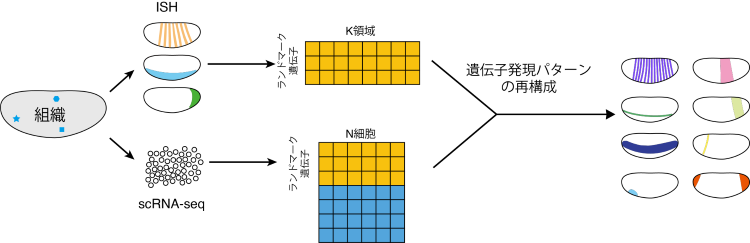
図1:空間的遺伝子発現パターンの再構成の流れ
【研究成果の内容】
本研究では、1細胞RNA-seq法およびISH法の特性の違いをモデル化することにより、両手法によって計測される遺伝子発現データ間の違いを補正する手法を開発しました。それに基づき、バラバラになった細胞の1細胞RNA-seqデータと位置情報を持つISHデータとを統合することが可能になりました。本手法ではまず、1細胞RNA-seqデータとISHデータとの類似度を測ることにより、1細胞RNA-seqデータの組織中での元の位置情報を推定します。そして、位置情報に基づいて1細胞RNA-seqデータを重み付け平均することで、全遺伝子の空間的遺伝子発現パターンを再構成します。研究グループは、この一連の計算プロセスをPerler(Probabilistic embryo reconstruction by linear evaluation of single-cell RNA-seq)と名付けました。
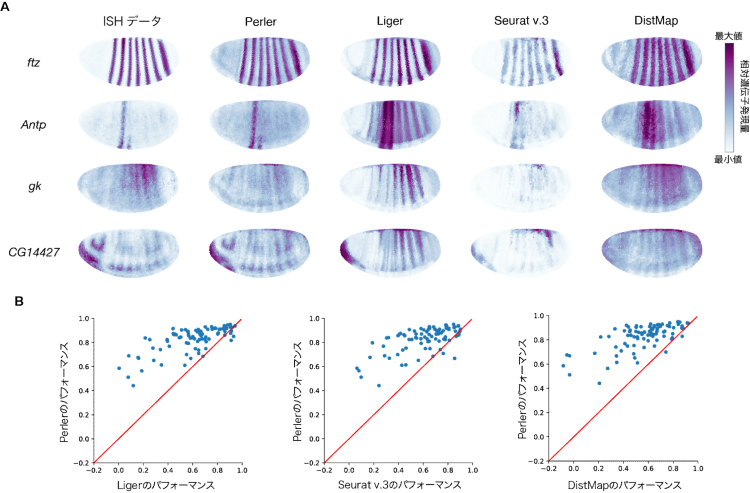
図2:再構成結果
(A)再構成された空間的遺伝子発現パターン(B)従来法との性能比較
Perlerをショウジョウバエ胚データに適用したところ、従来手法よりも高い精度で空間的遺伝子発現パターンを再構成することに成功しました(図2)。また、Perlerによる再構成をさらに詳しく調べたところ、一部の遺伝子にみられる特徴的な14本の周期的パターンの位相が正しく予測されていることがわかりました(図3)。このことは、1細胞の解像度でのPerlerの再構成の正確性を示すものであると考えられます。さらに、1細胞RNA-seqデータとISHデータの測定タイミングにずれが生じている場合でも、Perlerによる再構成は、ISHデータに過適合することなく、1細胞RNA-seqデータの時間情報を保持していることが示唆されました。Perlerは様々な組織(ゼブラフィッシュ胚、マウス肝臓、マウス大脳皮質)における遺伝子発現の空間パターンの再構成にも成功しており、このことはPerlerが、どの組織にも適用可能な一般的な手法であることを示しています。
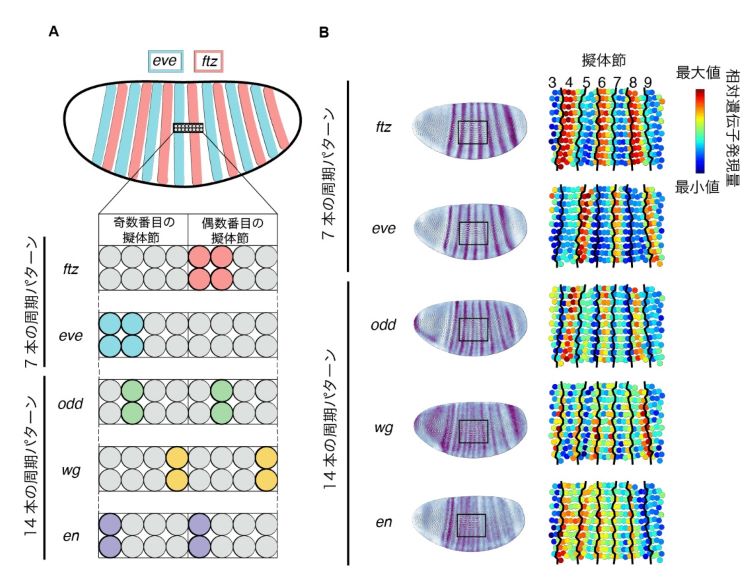
図3:1細胞解像度での再構成
(A)既知の空間的遺伝子発現パターン(B)Perlerによる再構成結果
【今後の展開】
本⼿法は遺伝子発現とその表現形である組織動態(生理機能や形作り)をつなぐ基盤技術を提供するものです。これまで様々な手法が同様の問題に取り組んできましたが、精度、一般性などの限界から、再構成された空間的遺伝子発現パターンに基づく研究が進んでいないのが現状でした。一方で、今回提案した機械学習法(Perler)は、従来法に比べて再構成精度を大幅に向上させることに成功し、また様々な組織に適用することが可能です。それにより、遺伝子一つ一つに対してISH実験を行うしかなかった状況から解放され、網羅的な空間的遺伝子発現パターンを利用した組織機能(生理機能や形作り)の研究が進むことが期待されます。例えば、再生医療として作成されたミニ臓器が、実際の体内臓器をどの程度再現しているのかなどの評価に応用されることが期待されます。
プロジェクトについて
本研究は、JST【ムーンショット型研究開発事業目標2050年までに、超早期に疾患の予測・予防をすることができる社会を実現】【JPMJMS2024-9】、自然科学研究機構生命創成探究センターExCELLS連携研究、日本学術振興会(JSPS)科学研究費助成事業の基盤研究(B)(No.21H03541、代表:本田直樹)、基盤研究(B)(No.17KT002、代表:近藤武史)、特別研究員奨励費(No. 20J23385、代表:坂口峻太)、内藤記念科学振興財団(代表:近藤武史)、京阪神グローバル研究リーダー育成コンソーシアム(K-CONNEX)(近藤武史)の支援を受けたものです。
論文情報
- 掲載誌: Nature Communications、2021
- 論文タイトル: Model-based prediction of spatial gene expression via generative linear mapping
- 著者名: Yasushi Okochi、Shunta Sakaguchi、Ken Nakae、Takefumi Kondo & Honda Naoki
- DOI: https://doi.org/10.1038/s41467-021-24014-x


 Home
Home









