広島大学大学院統合生命科学研究科 教授 坊農秀雅
Tel:082-424-4013
E-mail:bonohu*hiroshima-u.ac.jp
(*は半角@に置き換えてください)
本研究成果のポイント
- ゲノム編集メタデータベース(GEM)*1は、すべての生物種におけるゲノム編集情報を収集したデータベースであるが、機械的な情報収集ゆえ、どうしても複数のエラーが含まれてしまい、そのエラーは効率的かつ効果的なデータベース活用を阻害している。
- 本研究では、大規模言語モデル(LLM)*2を活用することで、機械的なエラー修正やデータ整備を行い、GEMの利用価値を高めることを検証した。結果、LLMの導入により、95.11%の精度で情報抽出が実行された。
- 本研究成果は、今後の遺伝子研究への貢献が期待される。
概要
広島大学大学院統合生命科学研究科の鈴木貴之大学院生と坊農秀雅教授は、大規模言語モデル(LLM)を活用した、ゲノム編集メタデータベース(GEM)の機械的なデータ整備の検証を行なった。GEMとは、PubMedと呼ばれる38,481,722件(2025年3月4日時点)の文献を収載する生命科学研究における最大規模の文献データベースから、ゲノム編集に関わる情報を機械的に抽出し公開しているデータベースである。今後のゲノム編集研究の促進への貢献を目指したデータセットと考える。
本研究では、GEMが抱える課題の1つである、収集した遺伝子情報のエラー、を機械的に修正し、GEM活用の幅を広げることを目的としている。LLMは、科学文献のような専門的な文章の文脈を把握した上で情報抽出するタスクに優れているという報告がされていることから、LLMの活用を検討及び検証した。
GEMの92,182データエントリーのうち、ヒトに関するデータ(266件のサブセット*3)について、LLMによる情報抽出タスクによるデータ整備を実行したところ、95.11%の精度で情報抽出が実行された。
本研究では、LLMの活用により、従来のGEMでは取得できていなかった情報を機械的に高精度で取得することで、GEMのデータ整備が可能であることが示された。本研究成果は、今後の遺伝子研究への貢献が期待される。
本研究結果は、国際学術雑誌「Database-The Journal of Biological Databases and Curation」に2025年3月8日付でオンライン掲載されました。
なお、本研究は広島大学から論文掲載料の助成を受けています。
発表論文
- 著者: Takayuki Suzuki1) and Hidemasa Bono1)2)*
*Corresponding author(責任著者)
1) 広島大学大学院統合生命科学研究科
2) 広島大学ゲノム編集イノベーションセンター - 論文タイトル: Pipeline to explore information on genome editing using large language models and genome editing meta-database
- 掲載誌: Database-The Journal of Biological Databases and Curation
- DOI:https://doi.org/10.1093/database/baaf022
背景
GEMは、ゲノム編集研究促進に向け、過去のゲノム編集研究に関する情報を網羅的かつ機械的に収集し公開している。しかし機械的な情報抽出手法上の問題により、データベース内にエラーが含まれることが課題の1つである。
研究成果の内容
GEMによって機械的に抽出された、ヒトに関する266件のサブセットについて、詳細にデータ内容をマニュアル(手動)で一つずつ調査することで、特に遺伝子情報についてどのような情報が含まれているかを確認した。この確認した情報を、基準(精度100%)とする。下記図の「1. Find related articles」では、266件のサブセット選抜方法及び、選抜されたサブセットに含まれる文献ID(PubMed ID)の取得方法を記載している。
マニュアル調査の結果、266件のサブセットには、大きく分けて4種類の遺伝子情報が含まれており、そのうちの2種類はエラー情報であることが明らかとなった。:ゲノム編集のターゲットとなった遺伝子(38.72%)、ゲノム編集によって発現変動したことが報告された遺伝子(24.44%)、ゲノム編集に関係のない文脈で記述されている遺伝子(10.53%)、抽出エラーによって収集された遺伝子(8.27%)。なお、現状のGEMでは、表示されている遺伝子情報が、4種類のうちのどの意味合いを持つデータであるかが不明確であることが課題である。
課題の解決に向け、文献内の文字列及びGEMの情報をもとに、LLMが「ゲノム編集のターゲットとなった遺伝子」を適切に選抜できるかを実験した。下記図の「2.Extract information from article」では、LLM活用方法の詳細を示している。実験の結果、LLMによる情報収集タスクの精度は95.11%となった。「ゲノム編集によって発現変動したことが報告された遺伝子」についても同様に実験を行い、その精度は85.28%であった。論文内では、LLMで抽出した情報の活用方法についても議論を行なっている。
今後の展開
本研究では、GEMのような専門分野のデータベースにおいて、LLMを活用することの有用性の一端を示唆した結果となったと考える。より詳細なゲノム編集に関わる遺伝子情報が検索可能となることで、今後のゲノム編集研究の有用な参考情報となることが期待される。
しかし、本研究ではヒト研究に関する266例のみで精度評価を行なったため、大規模なデータ、またはヒト以外の生物に関する研究文献において実行した場合の評価が今後は必要となると考える。また、遺伝子情報以外の専門的な情報、例えば、「実験のターゲットとなった生物種や細胞種」、「ゲノム編集による表現型」などの情報抽出におけるLLMの有用性の評価も今後の課題と考える。
参考資料
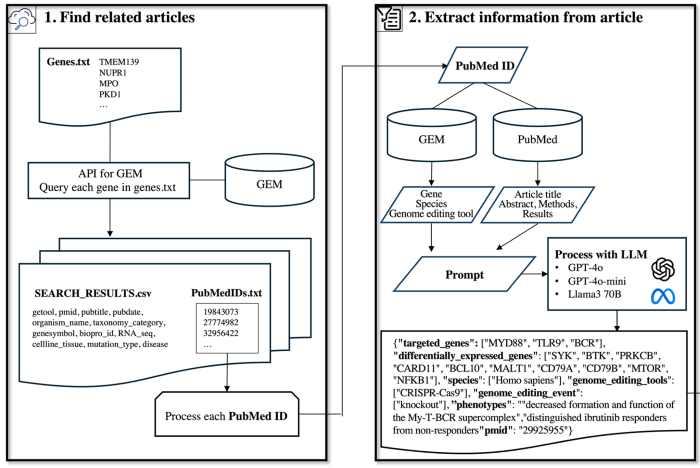
LLM活用によるデータ整備の概要図
用語解説
*1 ゲノム編集メタデータベース(GEM):PubMedデータベースや関連公共データベースの情報を基に、ゲノム編集研究に関する情報(メタデータ)を網羅的なデータセットにまとめて公開しているサービス。PubMed, PubMed Central, NCBI gene, PubTator, MeSH, NCBI taxonomy, EXTRACT 2.0の公共データベース及びサービスを活用することでデータ統合を実現している。49,048件の文献(2025年2月18日時点)において使用されたゲノム編集ツールと紐づいた遺伝子、生物種、細胞種、変異タイプ、疾患、関連IDを検索することができる。
具体的には、文献にて「ゲノム編集技術を使ってゲノムを編集した」という内容の記述がある場合に、どのようなゲノム編集実験をしているのかという情報を抽出している。
*2 大規模言語モデル(LLM):言語モデルとは、言語の予測による現実的な文章生成を目指した機械学習モデルを指す。大規模言語モデルは、ChatGPTやClaudeに代表される、大量のデータでトレーニングされた統計言語モデルとなっている。近年は様々な応用により、文章生成以外のタスク、例えば情報抽出など、様々な推論領域においてLLMの高精度なパフォーマンスが報告されている。
*3 サブセット:全体の一部分。ある集合体から、特定の要素を取り出した部分集合。


 Home
Home









