広島大学大学院統合生命科学研究科 教授 坊農 秀雅
Tel:082-424-4013
E-mail:bonohfu*hiroshima-u.ac.jp
(*は半角@に置き換えてください)
本研究成果のポイント
- ゲノム編集ツールの1つであるCBE(*1)が引き起こすRNA(*2)への意図しない編集(RNAオフターゲット作用)を網羅的に検出できるソフトウェアを開発しました。
- 人工知能(AI)の一種であるDNA言語モデル(*3)を活用し、RNAオフターゲット作用の予測精度を大幅に向上させました。
- 組織特異的な遺伝子発現(*4)データを用いて各組織でのリスクを予測するシステムを開発しました。
- RNAオフターゲット作用による生物へのリスクの予測に役立つAIを提供し、ゲノム医療の安全性向上に貢献することが期待されます。
概要
広島大学ゲノム編集(*5)イノベーションセンタープラチナバイオ共同研究講座の中前和恭共同研究講座助教と小野浩雅共同研究員(前任:ライフサイエンス統合データベースセンター特任助教)、広島大学大学院統合生命科学研究科の鈴木貴之大学院生(広島大学大学院リサーチフェロー)・米澤奏良大学院生(次世代AIフェロー)・坊農秀雅教授(プラチナバイオ共同研究講座教授・ライフサイエンス統合データベースセンター客員教授兼任)、株式会社ゲノムアナリティクスジャパンの山本謙太郎、角崎太郎(Independent Researcher)、ライフサイエンス統合データベースセンターの内藤雄樹客員准教授らはゲノム編集ツールの一種であるCBEが起こす意図しないRNA編集のリスクを分析するソフトウェアとリスク推定するためのAI予測ツールをDNA言語モデルを用いて開発しました。その結果、RNAオフターゲットを50%以上の正解率で予測することが確認され、本研究によってCBEを利用したゲノム医療の安全性評価の向上が期待されます。
関連論文等
論文タイトル:Risk Prediction of RNA Off-Targets of CRISPR Base Editors in Tissue-Specific Transcriptomes Using Language Models
掲載雑誌:International Journal of Molecular Sciences
DOI番号:https://doi.org/10.3390/ijms26041723
論文公開日:2025.2.18
著者:Kazuki Nakamae※1, Takayuki Suzuki2, Sora Yonezawa2, Kentaro
Yamamoto3, Taro Kakuzaki4, Hiromasa Ono1,5, Yuki Naito5,
Hidemasa Bono1,2,5 ※責任著者
著者所属:1 広島大学ゲノム編集イノベーションセンター
2 広島大学大学院統合生命科学研究科
3 株式会社ゲノムアナリティクスジャパン
4 Independent Researcher
背景
CBEはCRISPR-Casシステム(*6)を利用して狙ったDNA領域にシトシン(C)→チミン(T)塩基置換(*7)を導入することができるゲノム編集ツールであり、ゲノム上にDNA二本鎖切断(*8)を起こさずに遺伝子を改変できる画期的な技術です。この技術を利用することで遺伝子疾患の原因の一つとなる一塩基多型(SNP)を修正できる可能性があり、ゲノム医療への活用が期待されています。その一方で、CBEには細胞中のRNA分子に作用し、意図しないシトシン(C)→ウラシル(U)置換を導入する現象(RNAオフターゲット作用)が知られており、細胞への毒性が表出するなど安全性において懸念もあります。CBEのRNAオフターゲット作用はRNA上のACW塩基モチーフ (Wはアデニン(A)もしくはチミン(T)/ウラシル(U)を指す)上で発生しやすいということが先行研究で報告されていましたが、その塩基モチーフに当てはまらない非標準的な(Non-canonical)RNAオフターゲット作用については詳しく調べられておらず、分析手法そのものも標準化されていませんでした。
研究成果の内容
本研究ではまずRNAシーケンスデータからRNAオフターゲット作用を分析するためのソフトウェア「Pipeline for CRISPR-induced Transcriptome-wide Unintended RNA Editing:PiCTURE」を開発しました。そしてCBE導入ヒト培養細胞HEK293TサンプルとCBE非導入ヒト培養細胞HEK293Tサンプル(Cas9ニッカーゼ導入サンプル)由来のRNAデータに対して「PiCTURE」による分析を実施し、CBE導入サンプルでのみ観測されたC→U置換をCBE特異的なRNAオフターゲット領域として同定した上で、基質周辺にACWモチーフを持つ領域をCanonical RNAオフターゲット、ACWモチーフを持つ部位のない領域をNon-canonical RNAオフターゲットとして分類しました。(図1A)。例えば、CBEの一種であるBE4-rAPOBEC1を導入したサンプル群ではRNAオフターゲット領域232,254箇所のうち、119,359(51.4%)箇所がCanonical RNAオフターゲットであり残り48.6%の箇所はNon-canonical RNAオフターゲットでした。このことからACW塩基モチーフに従わないRNAオフターゲットも多数存在しえることが示されました(図1B左側棒グラフ)。また、PiCTUREのモチーフ解析結果から示唆されたWCW塩基モチーフをRNAオフターゲット領域232,254箇所に適用した場合は、159,545(68.7%)箇所がWCWモチーフをもつということが分かり(図1B右側棒グラフ)、WCW塩基モチーフもRNAオフターゲットを観測する上で有用であることが示されました。
次にRNAオフターゲットを配列から予測するモデルの構築を目指しました。モデル構築において、著者らは様々な生物に由来する塩基配列(累計約324億9千塩基)を学習しDNA言語モデルとして公開されている「DNABERT-2」事前学習モデルに着目し、「PiCTURE」で解析したCBE特異的RNAオフターゲット配列でファインチューニングを実施することでCBE特異的RNAオフターゲットを予測する機械学習モデル「STL(別称:RNAOffScan v1)」と「SNL(別称:RNAOffScan v2)」を開発しました(図1C)。「STL」と「SNL」のRNAオフターゲットの検出性能を評価したところ、Canonical RNAオフターゲットとNon-canonical RNAオフターゲットの検出のいずれにおいても正解率(Accuracy)が50%を超えており, 適合率(Prediction), 再現率(recall), F1スコアの4つの精度指標において、ACWまたはWCW塩基モチーフでの検出の性能を有意に超えることを示しました。
最後にこのようなRNAオフターゲット作用のAI予測ツールの活用例として、CBEのRNAオフターゲットリスクを人体の組織や器官ごとに推定する新規ソフトウェア「Predicting RNA Off-target compared with Tissue-specific Expression for Caring for Tissue and Organ:PROTECTiO」を構築しました。「PROTECTiO」は、組織特異的な遺伝子発現プロファイルを提供しているDBCLSの「RefEx」データベースと連携しています。RefExから得られた組織特異的に発現上昇している遺伝子のトランスクリプト配列に対して、C→U変換によってCDS領域に終止コドンが出現するシトシン塩基(Potential Rrsk Substrates; PRSs)をスクリーニングします。その上で、RNAオフターゲット予測モデルによるPRSsのリスク判定とリスクと判定されたシトシン塩基の密度(Effective Substrate Density:ESD)を算出した上で、ESDの合算値を組織特異的なRNAオフターゲットリスクとしています。このような「PROTECTiO」の算出アルゴリズムをヒトデータに適用すると、脳や卵巣ではCBE特異的なRNAオフターゲットリスクが相対的に低いと予測された一方で、結腸や肺では高いリスクがあることが予測されました。
本ソフトウェアは商用利用問わず誰でも無償利用できるコードとして公開されています。
• PiCTURE:https://github.com/KazukiNakamae/PiCTURE
• RNAOffScan v1:https://huggingface.co/KazukiNakamae/STLmodel
• RNAOffScan v2:https://huggingface.co/KazukiNakamae/SNLmodel
• PROTECTiO:https://github.com/KazukiNakamae/PROTECTiO
今後の展開
本研究で開発されたRNAオフターゲット作用のAI予測ツールが、将来のゲノム編集研究の場で活用され、安全性の事前予測が可能になるとともにさまざまな実験においける本AI予測の可用性が検証されていくことが期待されます。また、本研究で提供されたRNAオフターゲット作用の分析と予測の標準的な枠組みは、CBEの安全性評価において先例のない視点とアプローチを提供しており、ゲノム編集ツールの改良による特異性の向上とより安全なゲノム治療技術の検討が促進されることが期待されます。
参考資料
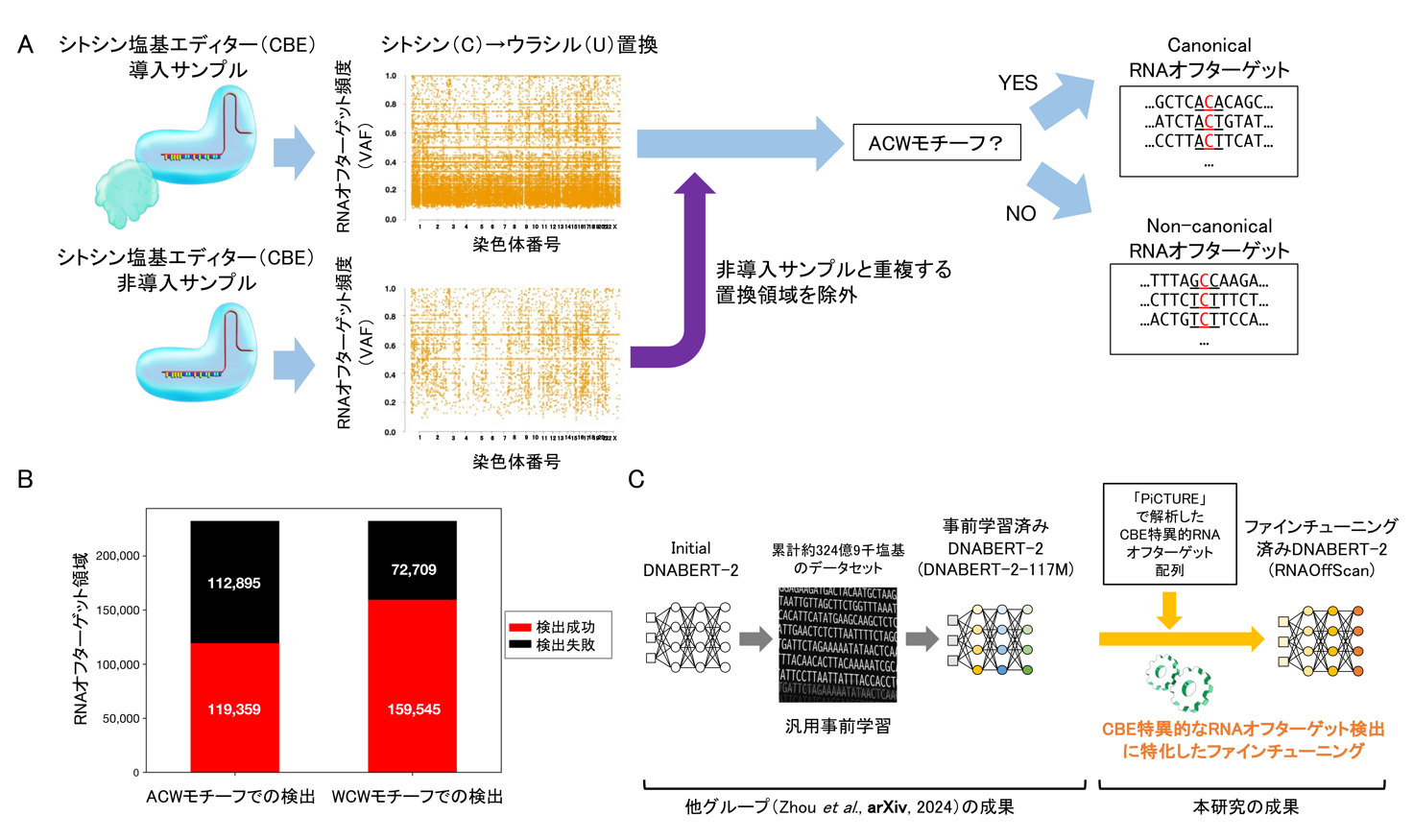
図1 CBE特異的なRNAオフターゲットの同定とDNA言語モデルのファインチィーニングの概略図。
(A)CBEを導入したサンプルとCBEを導入せずに代わりにCas9ニッカーゼを導入したサンプルからシトシン(C)→ウラシル(U)置換を検出しています。検出した置換領域についてRNAオフターゲット頻度とRNAの発現元となった染色体位置をみると広範に置換が生じていることがわかります。これらのデータに対してCBE導入サンプルとCBE非導入サンプルの置換領域が重複している領域を除外したRNA配列領域をCBE特異的なRNAオフターゲット領域として同定しました。RNAオフターゲット領域については基質周辺にACWモチーフを持つ領域をCanonical RNAオフターゲット、ACWモチーフを持つ部位のない領域をNon-canonical RNAオフターゲットとして分類しています。(B)同定したRNAオフターゲット領域について基質周辺にACWモチーフあるいはWCWモチーフで検出可能な領域と領域とそうでない領域をそれぞれカウントしました。(C)モデル構築において、他グループ(Zhou et al., arXiv, 2024)がInitial DNABERT-2に対して汎用事前学習として累計約324億9千塩基のデータセットを与えて構築した事前学習済みDNABERT-2(DNABERT-2-117M)に対して、図1Aで示したようなCBE特異的RNAオフターゲット配列と非特異的RNAオフターゲット配列データをファインチューニング用データとして入力しています。その結果として、CBE特異的なRNAオフターゲット検出に特化したファインチューニング済みDNABERT-2(RNAOffScan v1/v2)を構築することができました。
用語解説
*1 CBE:正式名称は「シトシン塩基エディター(Cytocine Base Editor)」。CRISPR-Casシステムを利用して狙ったDNA領域にシトシン(C)→チミン(T)塩基置換を導入することができるゲノム編集ツールの一つ。
*2 RNA:正式名称は「リボ核酸(Ribonucleic acid)」。リボヌクレオチドという物質が一本鎖状に重合した物質。アデニン(A)、グアニン(G)、シトシン(C)、ウラシル(U) という4つの「塩基」から構成されている。細胞内ではゲノムDNAの情報を外部に伝える媒体として合成され、タンパク質はRNAに基づいて合成されます。このような利用形態下にあるRNAを特にメッセンジャーRNA(mRNA)と呼称します。
*3 DNA言語モデル:生物の遺伝情報を記したDNA塩基配列をコンピュータに読み取らせて、人間の言葉の文章のようなパターンや意味として解釈できることを期待して構築されたAIシステムを指します。このシステムを利用することでDNAの中で特に重要な働きをする部分や見つけたり、DNAの一部が変化したときにそれが生物にどんな影響を与えるかを予測したりすることができます。
*4 発現:ゲノムDNA内の遺伝情報が細胞における構造および機能に反映されるプロセスを指す。具体的には、ゲノムDNAの塩基配列に基づいてRNAおよびタンパク質が合成されるプロセスを指す。
*5 ゲノム編集
細胞内にある生物の遺伝情報(ゲノム)上に存在するDNA配列に対してヌクレアーゼ(DNA 切断酵素)等を作用させることで遺伝子が改変する技術です。
*6 CRISPR-Casシステム
原核生物のもつ獲得免疫システムの一つで、核酸等の外来物質の分解を行うことができる。CRISPR-Cas酵素は6つのType(I–VI型)に分類されます。Type II CRISPR-Cas酵素であるCas9ヌクレアーゼはガイドRNAとの複合体を形成し、ガイド配列と相補的な二本鎖DNAを部位特異的に切断することができるため、様々な生物でのゲノム編集に利用されています。
*7 塩基置換:DNAやRNAを構成する塩基(アデニン(A)、グアニン(G)、シトシン(C)、チミン(T)、ウラシル(U))が別の種類の塩基に変換することです。
*8 DNA二本鎖切断:細胞内のDNAは鎖状の2本がらせん状に対合していますが、その両方の鎖が同時に切断される現象です。過剰な切断が起こると遺伝子情報や細胞によくない影響を与えることが知られています。

 Home
Home



