○九州大学大学院農学研究院 准教授
中村 崇裕(なかむら たかひろ)
電話・FAX:092-642-3370
Mail:tnaka*agr.kyushu-u.ac.jp
○広島大学大学院理学研究科 教授
山本 卓(やまもと たかし)
電話:082-424-7446
FAX:082-424-7498
Mail:tybig*hiroshima-u.ac.jp
Mailの*は半角@に置き換えて送信してください。
平成25年4月24日
国立大学法人 九州大学
国立大学法人 広島大学
PPR蛋白質の核酸認識コードの解読に成功
―任意のRNAまたはDNA配列に結合する蛋白質の開発が可能に―
九州大学大学院農学研究院の中村崇裕准教授は、医学研究院の大川恭行准教授、広島大学大学院理学研究科の山本卓教授との共同研究で、PPR蛋白質のRNAおよびDNA認識コードの解明に成功しました。
RNAまたはDNAと配列特異的に結合するPPR蛋白質は35アミノ酸の「PPRモチーフ(※1)」十数個の連続で構成されています。今回の共同研究により、以下のことを発見しました。
・PPRモチーフと塩基が1対1で対応すること。
・連続したPPRモチーフが核酸配列中の連続した塩基を認識すること。
・モチーフ中の3箇所のアミノ酸で結合塩基を指定すること。
・結合塩基を予測、設計可能であること。
・DNAまたはRNAの両方に適用可能であること。
このPPR蛋白質の核酸認識コードの解読により、任意のRNAまたはDNA配列に結合する蛋白質の開発が可能になりました。今後、医療、農林水産業などの様々な生物系産業への利用が期待されます。
なお、RNA結合型PPR蛋白質に関しては、PCT/JP2012/077274(平成24年10月22日出願)、DNA結合型PPR蛋白質に関しては、特願2013-89840(平成25年4月22日出願)として、特許を出願しました。
様々な生物のゲノム情報が明らかになり、その利用による研究技術の開発、医療への応用、有用な農作物の開発などが盛んに行われています。しかし、多くの生物において、ゲノム中の目的遺伝子のみを選択的に破壊するジーンターゲッティング法(※2)は確立しておらず、ゲノム情報の利用に大きな制限がかかっていました。
近年、設計可能な人工制限酵素(人工ヌクレアーゼ(※3))を用いて、ゲノム上の特定の遺伝子を破壊したり、標識遺伝子を導入したりするゲノム編集技術が注目されています。同一の技術体系によって、植物、微生物、動物を含む全ての生物種のゲノムが改変できるため、基礎研究および様々な生物系産業での利用が期待されています。ゲノム編集には核酸と配列特異的に結合する核酸結合モジュール(※4)が必須です。現在、DNA結合モジュールとして、ジンクフィンガー、TALE、CRIPSR/Cas(※5)が利用されており、それぞれの核酸結合モジュールの比較検討が進められています。しかし、ゲノム編集技術はいまだ萌芽期であり、新たな核酸結合モジュールの発見が期待されています。
一方、最近、様々な生命現象におけるRNAの重要性が認識されてきました。例えば、低分子のRNA成分を利用したRNA干渉法(※6)による目的遺伝子転写物の分解(ノックダウン)技術が盛んに利用されています。しかし、RNAに作用する蛋白質性結合モジュールは、未だ産業化されていないのが現状です。
PPR(pentatricopeptide repeat)蛋白質は植物で大きなファミリー(※7)を形成する蛋白質です。それぞれが異なる配列に作用するDNAまたはRNA結合蛋白質として働くことが知られています。
研究グループは、これまでに研究されてきたRNA結合に働く数十個のPPR蛋白質とその結合配列のデータを基にしたコンピュータ解析により、PPR蛋白質のRNA認識コードを解読しました。
その結果、PPRモチーフとRNA塩基が一対一の対応関係で結合すること、特定の3箇所のアミノ酸の組み合わせで結合塩基が決定すること、結合塩基がプログラム化可能であること、を明らかにしました(図1)。また、解読したRNA結合コードがDNA結合に働くPPR蛋白質にも適用できることを見出しました。
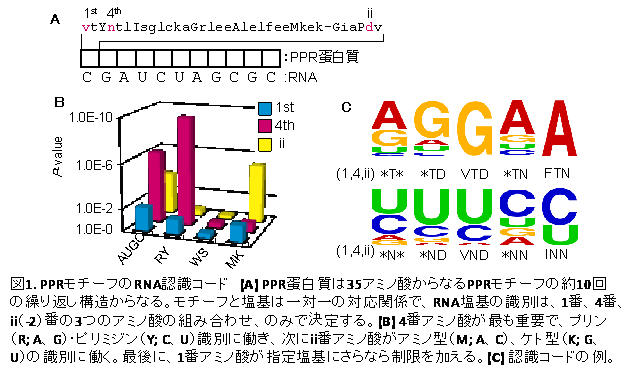
PPR蛋白質の核酸認識コードの解明により、意図するRNAまたはDNA配列に特異的に結合する蛋白質を開発できるようになりました。また、生物にとって重要なPPR蛋白質、産業に有用な天然型PPR蛋白質の標的配列の予測も可能になりました。
PPR蛋白質は、DNAまたはRNAの両方の核酸に対応可能なモジュールであり、既知のゲノム編集用核酸結合モジュールにない特徴の1つです。また、TALEで知られるTを認識するゼロモチーフのようなものはPPRには見当たらず、完全に自由な配列選択性を有しています。
DNAを標的としたゲノム編集は、国際的に大きな注目を集めている新技術です。DNA結合型PPRを核酸結合モジュールに用いた日本発のゲノム編集技術の創出が期待されます。
RNA結合型PPRを用いて、RNAを切断したり、特定の塩基を置換する編集技術、特定のRNAの局在を見る可視化技術などの革新的なRNA操作技術の創出が期待されます。
*本研究成果の一部は、農業・食品産業技術総合研究機構 生物系特定産業技術研究支援センターが実施するイノベーション創出基礎的研究推進事業の研究課題「植物ミトコンドリア遺伝子発現の分子基盤解明と育種への応用」(平成21〜23年度)として行ったものです。
(※1)モチーフ
繰り返し単位のこと。タンパク質においては、一定規則の複数個のアミノ酸から構成される機能的または構造的なひとまとまりのこと。
(※2)ジーンターゲッティング法
分子生物学の実験で、特定の遺伝子に変異を導入したり組換えたりする技術のこと。狙った遺伝子だけに効率よく変異を導入できることが特徴。
(※3)人工ヌクレアーゼ
特定のDNAに結合するドメインとDNA切断酵素を結合させた人工タンパク質。これまで遺伝子工学で使われていた制限酵素は約2000塩基対から一カ所を選択的に切断することができたが、人工ヌクレアーゼを使えば、30億塩基対あるヒトゲノムから一カ所を選択的に切断することができる。
(※4)モジュール
機能単位、交換可能な構成部分という意味。複数のモジュールを組み合わせることで、複数の機能を付与することができる。
(※5)ジンクフィンガー、TALE、CRIPSR/Cas
それぞれ、人工ヌクレアーゼを作製する際に使用されているDNAに結合するドメイン、もしくは遺伝子座の名称。これらを使うことで、任意のDNA塩基配列を認識するタンパク質、もしくはタンパク質複合体を作製することができる。
(※6)RNA干渉法
二本鎖RNAと相補的な塩基配列を持つmRNAが分解されるRNA干渉(RNA interference、RNAi)という現象を利用して、人工的に二本鎖RNAを導入することにより任意の遺伝子の発現を抑制する手法。
(※7)ファミリー
タンパク質においては、進化上の共通祖先に由来すると推定されるタンパク質をまとめたグループのことを指す。似たような機能、構造を持つ場合が多い。
○九州大学大学院農学研究院 准教授
中村 崇裕(なかむら たかひろ)
電話・FAX:092-642-3370
Mail:tnaka*agr.kyushu-u.ac.jp
○広島大学大学院理学研究科 教授
山本 卓(やまもと たかし)
電話:082-424-7446
FAX:082-424-7498
Mail:tybig*hiroshima-u.ac.jp
Mailの*は半角@に置き換えて送信してください。
○九州大学 広報室
〒812-8581福岡市東区箱崎6-10-1
TEL:092-642-2106
FAX:092-642-2113
Mail:koho*jimu.kyushu-u.ac.jp
URL:http://www.kyushu-u.ac.jp
○広島大学 学術・社会産学連携室広報グループ
〒739-8511 東広島市鏡山一丁目3-2
TEL:082-424-6762
FAX:082-424-6040
Mail:koho*office.hiroshima-u.ac.jp
URL:http://www.hiroshima-u.ac.jp/index-j.html
Mailの*は半角@に置き換えて送信してください。
Copyright © 2003- 広島大学