<報道に関すること>
公益財団法人かずさDNA研究所 広報・研究推進グループ
TEL: 0438-52-3930
<研究に関すること>
公益財団法人かずさDNA研究所
植物ゲノム・遺伝学研究室 室長 磯部 祥子
TEL: 0438-52-3935
TEL: 0438-52-3930
※2月4日と5日は担当者不在のため、広報・研究推進グループにて取次いたします。
【用語解説】
*1 遺伝子:
親から子へと遺伝する、あるいは細胞から細胞へと伝えられる形質を決定する因子であり、生物の体を作り動かすのに必要なタンパク質などを作るための設計図のことで、その本体はDNAである。
*2 ゲノム:
生物をその生物たらしめるのに必須な最小限の染色体のひとまとまり、またはDNA全体のことをいう。
キク科に属する植物種は被子植物の約10%を占め、特に栽培ギクの起源となったキク属は主に東アジアで分化したことから、植物の多様性を研究する上で重要な植物群のひとつです。また、栽培ギクは日本の切花生産の約40%を占めており、世界的にもバラやカーネーションと並んで重要な花き品目です。
今回解析したキクタニギク(Chrysanthemum seticuspe)は、キク科キク属の多年草で、東北から九州にかけて自生しており、10~11月に黄色い花をつけます。キクタニギクの染色体数は2n=18ですが、栽培されているキクは多くの野生ギクが交雑を繰り返し品種として成立したため、複雑なゲノム構造になっています。そのため、ゲノム構造が単純なキクタニギクが、キクのモデル植物として開花制御の研究などに利用されています。
キク属の遺伝学的研究がこれまで進んでいなかった理由としては、自家不和合性もあります。通常のキクタニギクは、同じ個体の花にある雄蕊(ゆうずい;おしべ)の花粉が雌蕊(しずい;めしべ)についても種子がほとんどできません(他殖性、と言います)。そのため、遺伝学的な研究に重要な、ゲノム構造が同質で安定した形質が現れる、いわゆる「純系」と呼ばれる系統をつくることができなかったのです。
広島大学大学院理学研究科の谷口特任准教授と草場信教授は、同一個体の花粉で種子を作ることができる(自殖性)個体「AEV2」を発見し、文部科学省・ナショナルバイオリースプログラムのもと、この「AEV2」を用いて5回の自殖を繰り返しました。そして、ゲノム配列の構造がより同質化された系統「XMRS10」を作出し、ゲノム解析を行いました。
本研究では、広島大学で作出された「XMRS10」を用いて、かずさDNA研究所および農研機構野菜花き研究部門でゲノム配列の解読と遺伝子予測、連鎖地図の作成を行いました。また、また、農研機構野菜花き研究部門と東京大学、日本大学において生育・開花に関わる遺伝子の探索を実施しました。キクタニギクのゲノムサイズの推定は農研機構野菜花き研究部門で、栽培キクゲノムの配列変異の検出はかずさDNA研究所で行いました。
キクタニギクは、文部科学省・ナショナルバイオリソースプロジェクト(NBRP)広義キク属で、バイオリソースとして扱われています。プロジェクトについては、こちらをご覧ください。
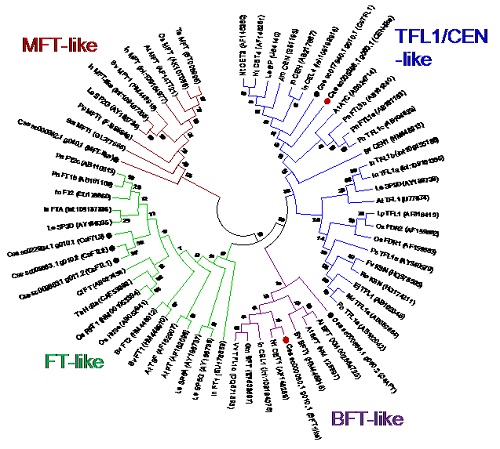
探索した開花に関連する遺伝子のうち、花成ホルモン(フロリゲン)を含むPEBP (phosphatidylethanolamine-binding protein) ファミリーに属するに関連する遺伝子群と他種で同定されている遺伝子との配列の類似性を示した系統樹。黒丸は既報のキクタニギク遺伝子。赤丸はゲノム解読により初めて推定された遺伝子。
<報道に関すること>
公益財団法人かずさDNA研究所 広報・研究推進グループ
TEL: 0438-52-3930
<研究に関すること>
公益財団法人かずさDNA研究所
植物ゲノム・遺伝学研究室 室長 磯部 祥子
TEL: 0438-52-3935
TEL: 0438-52-3930
※2月4日と5日は担当者不在のため、広報・研究推進グループにて取次いたします。
掲載日 : 2019年02月04日
Copyright © 2003- 広島大学