広島大学大学院理学研究科・クロマチン動態数理研究拠点の新海創也特任助教らの研究グループは、ヒトの生細胞の核内クロマチンの動きを表現する数理モデルを考案しました。実際に、その動きを精密に観測したところ、遅くて異常な動きはクロマチンドメインの構造に拘束された動きであることを突き止めました。また、生きている状態のクロマチンドメインの大きさとフラクタル次元(注3)といった構造情報を得ることに成功しました。
本研究成果は、「PLOS Computational Biology」オンライン版に10月20日(米国東部標準時間)に掲載されました。
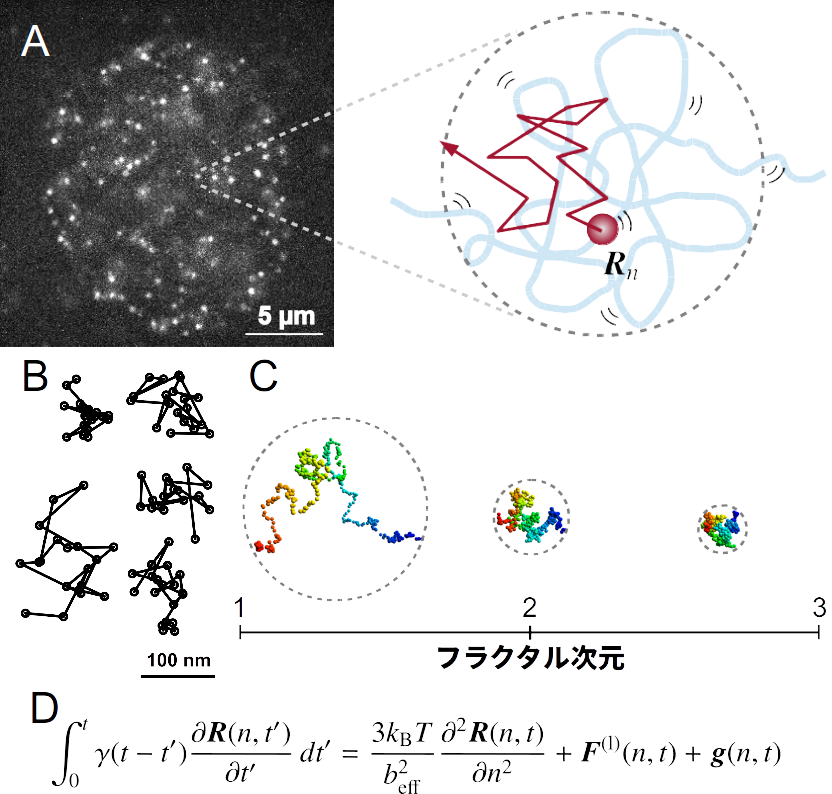
(A)核内のDNAにつけた蛍光分子が自発的にランダムに光っている様子をとらえた顕微鏡画像(左)。最近の染色体構造捕獲法の知見を踏まえると、画像の輝点はクロマチンドメイン内の一つのヌクレオソームに対応すると考えられる(右)。0.05秒毎に画像を取得することで、その輝点が動いていることがわかった。しかしながら、クロマチンドメインの全貌はわからない。(B)約1秒間での典型的なヌクレオソームの軌道が5つ描かれている。100ナノメートルの領域をランダムに動く。(C)クロマチンドメインの疎密の度合いは1~3の間の値をもつフラクタル次元を使って特徴づけることができる。(D)核内ヌクレオソームの動きを説明する数式。
【本研究成果のポイント】
- 細胞の核内で折りたたまれたひも状のDNAを表現する数理モデルを考案し、その動きと構造(ひもの形)の関係を表す理論式を導き出した。
- ヒトの生細胞でDNAにつけた蛍光分子の動きを精密に計測し、上の理論式を用いることで、生きている状態でのDNAの構造情報を得ることに成功した。
- 本研究での数理モデルに基づく解析を展開することで、クロマチンの動きと対応する遺伝子発現調節機構の関係の解明につながり、数理科学的アプローチによる遺伝子発現の予測や創薬への応用に役立つと期待される。
【論文に関する情報】
- DOI番号:10.1371/journal.pcbi.1005136
- 論文題目:Dynamic Nucleosome Movement Provides Structural Information of Topological Chromatin Domains in Living Human Cells
- 著者:Soya Shinkai*, Tadasu Nozaki, Kazuhiro Maeshima and Yuichi Togashi
*Corresponding author(責任著者)
【本研究成果に関する問い合わせ先】
大学院理学研究科・クロマチン動態数理研究拠点 特任助教 新海 創也
Tel:082-424-5529
E-mail:soya*hiroshima-u.ac.jp (*は半角@に置き換えて送信してください)

 Home
Home