大学院統合生命科学研究科
特任教授 坊農 秀雅
Tel:082-424-4013
E-mail:bonohu*hiroshima-u.ac.jp (注: *は半角@に置き換えてください)
本研究成果のポイント
- 低酸素刺激によって発現変動する遺伝子は低酸素誘導因子に代表される低酸素応答システムの発見に伴いよく研究されていますが、有名な遺伝子の影に隠れて、まだ明らかになっていない低酸素応答性遺伝子があるのではないかと考えました。
- そこで本研究では、公共データベース(公共DB)に蓄積したデータを自らの研究に利用するバイオデジタルトランスフォーメーション(バイオDX)を活用して、新規の低酸素応答性遺伝子探索をデータ駆動的に試みました。
- 公共DBから収集した低酸素刺激前後のすべての遺伝子の発現量の変化を評価するメタ解析手法を構築し、論文データベースにあるすべての遺伝子の情報を統合するマルチオミックス解析によって、新規の低酸素応答性遺伝子を複数発見しました。
概要
広島大学大学院統合生命科学研究科の小野擁子大学院生と坊農秀雅特任教授は、複数の公共データベース(公共DB)を統合するマルチオミックス解析によって新規の低酸素応答性遺伝子を発見しました。
低酸素刺激によって発現変動する遺伝子は、低酸素誘導因子1(HIF-1)に代表される低酸素応答システムの発見など、活発に研究されています。よく研究されている分野は出版バイアスの影響をより強く受けてしまうため、例えば血管内皮増殖因子Aなどの古くから知られている遺伝子であるほど研究対象になります。そのため私たちは、この出版バイアスの影に隠れている未知の低酸素応答性遺伝子があるのではないかと考えました。
現在、公共DBに蓄積したデータを自らの研究に利用するバイオデジタルトランスフォーメーション(バイオDX)が注目されています。この公共DBを活用することで、データ駆動的に新規の低酸素応答性遺伝子の探索を試みました。
公共DBから収集した低酸素刺激有無のすべての遺伝子の発現情報(トランスクリプトーム)の変化を評価する、データ駆動型のメタ解析手法を構築しました。収集したトランスクリプトーム情報と、公共DBにあるすべての論文(ビブリオーム)に掲載された遺伝子の情報を統合するマルチオミックス解析によって、新規の低酸素応答性遺伝子を複数発見しました。
今回の研究成果を元に有用物質生産生物のゲノム編集に向けて、開発したマルチオミックス解析手法を応用していくことが期待されます。また、解析の結果発見した、低酸素刺激によって発現が低下する遺伝子群と非コードRNAの代謝のかかわりを今後検討していく予定です。
本研究成果は、スイスの出版社Multidisciplinary Digital Publishing Institute(MDPI)のBiomedicines誌に掲載されました。
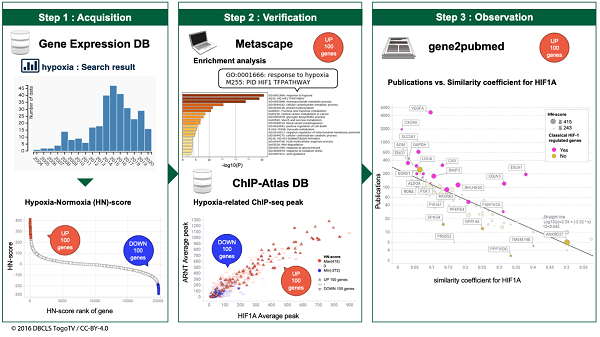
図1 マルチオミックス低酸素トランスクリプトームメタ解析
ステップ1.低酸素応答性遺伝子の公共DBからの抽出。ステップ2.既知の低酸素刺激関連遺伝子を用いたデータセットの評価。ステップ3.低酸素刺激応答に関連する新規遺伝子の発見。
論文情報
- 掲載誌: Biomedicines
- 論文タイトル: Multi-omic meta-analysis of transcriptomes and the bibliome uncovers novel hypoxia-inducible genes
- 著者名: Yoko Ono, Hidemasa Bono* *Corresponding author(責任著者)
- DOI: https://doi.org/10.3390/biomedicines9050582

 Home
Home