<研究に関すること>
広島大学大学院統合生命科学研究科
教授 草場 信
Tel:082-424-7490 FAX:082-424-0738
E-mail:akusaba*hiroshima-u.ac.jp
<広報に関すること>
広島大学財務・総務室広報部広報グループ
Tel:082-424-3749 FAX:082-424-6040
E-mail:koho*office.hiroshima-u.ac.jp
(注: *は半角@に置き換えてください)
いわゆる「菊」は栽培ギクと呼ばれ、ゲノムを6セットもつ六倍体であるうえ、自家不和合性(*4)であるためヘテロ性(*5)が高く、そのゲノム構造は非常に複雑です。そのため、栽培ギクで遺伝学的研究を行うのは難しいことが知られています。その問題を克服するために、栽培ギクに良く似た性質を持つ二倍体種キクタニギクの純系化系統Gojo-0がモデル系統として開発されています。今回、広島大学大学院統合生命科学研究科の草場信教授(専門:植物分子遺伝学)らの共同研究チームは、このGojo-0系統の全ゲノム塩基配列についてロングリードシーケンスを用いることにより高精度・染色体レベルで決定することに成功しました。
本研究成果は、10月7日に科学誌「Communications Biology」にオンライン掲載されました。
【背景】
栽培ギクは世界三大花きにも数えられる、日本の切り花生産の1/3を占める産業的に重要な花き品目です。栽培ギクの花は江戸時代以前からの長い間の品種改良の結果、非常に多様な形と色を持っており(図1)、その品種分化の解明は非常に興味深いものがあります。
栽培ギクは六倍体であるなど、非常に複雑なゲノム構成を持っているため、遺伝学的な解析が難しく、研究が進んでいませんでした。そこで栽培ギクに性質がよく似た二倍体野生ギク・キクタニギクを用いて研究が進められ、特に「電照ギク」で知られている開花の日長制御メカニズムの解明において大きな成果が挙げられています。
一方、全ゲノム塩基配列は分子レベルでの解析に非常に有用であることから、私たちはこれまでにキクタニギクのドラフトシーケンスを得て、公表しています。今回は、純系化され、モデル系統として確立されているGojo-0系統を用い、染色体レベルの高精度全ゲノム塩基配列を取得し、その解析を行いました。染色体レベルの全ゲノム塩基配列の報告は、キク属だけでなくキク連においても初めてです。
【研究成果の内容】
・HiSeq(*6)によるショートリードシーケンス・PacBio sequel(*7)によるロングリードシーケンスとHi-C法(*8)を用いた解析により、キクタニギク(2n=18)の9本の染色体に対応する巨大配列を得ました。キクタニギクのゲノムサイズは3.15Gbですが、取得した全配列はその97%、染色体に位置付けられた配列は95%であり、ほぼゲノム全体を網羅した染色体レベルの配列を明らかにしたことになります。
・推定された遺伝子数は約74,000と多く、これはモデル植物として有名なシロイヌナズナの約2.7倍でした。遺伝子数の拡大はゲノム重複が進化の過程で起きた影響であることが、解読された配列からも推定されました(図2)。
・キクタニギクのゲノムの80%は反復配列であることが分かりました。また、そのうちの多くは‘動く遺伝子’レトロトランスポゾンでした。
・染色体レベルのゲノム配列を用いることで、突然変異体の原因遺伝子の単離が格段に容易になることを示しました。これはこれまでのドラフトシーケンス(*9)ではできなかったことです。
・花の形態を制御する遺伝子群の解析などから、六倍体である栽培ギクのゲノムと二倍体であるキクタニギクのゲノムに非常に良い対応があることが確かめられました。また、ゲノムの構成も非常によく対応していることがわかり(図3)、キクタニギクゲノム配列が栽培ギク研究の非常に良い参考になることが明らかになりました。今後、栽培ギクの品種改良にも生かされていくことが期待されます。
・キクタニギクの花形成の突然変異体shiboridamaでは、花形成のマスター制御遺伝子であるLEAFY(CsFL)(*10)に新発見のレトロトランスポゾンSbdRTが挿入されていることが分かりました(図4)。SbdRTは染色体上の遺伝子が多く含まれる領域に多く、遺伝子周辺に転移しやすいことが推察されました(図5)。

図1 様々な花の形態・花色を持つ栽培ギク
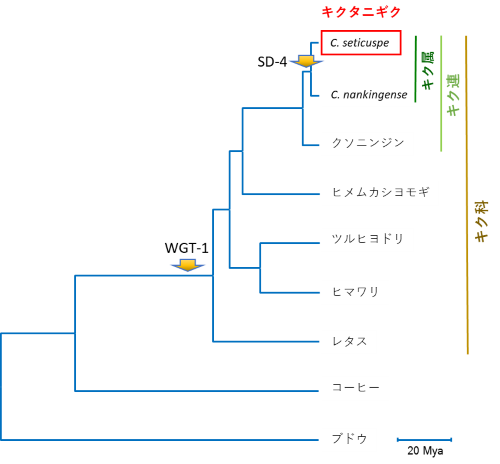
図2 全ゲノム塩基配列を用いて作成された分子系統樹。進化の過程で、キクタニギクはキク科に共通する全ゲノム重複(WGT-1)の他、キク属に特有の部分的ゲノム重複(SD-4)を経験していると推定されました。

図3 キクタニギクと栽培ギクの遺伝子の染色体上の位置関係。9本の連鎖群上の遺伝子の位置関係がよく対応していることがわかる。栽培ギクのゲノムは同質六倍体に近い構成であるため、栽培ギクの遺伝子も9本の連鎖群に収束している。

図4 キクタニギク(左)とshiboridama変異体(中)。 shiboridamaでは頭状花序(*11)のひとつひとつの花の器官が全て葉様組織に転換している。shiboridamaのCsFLには約8kbのSbdRTの挿入があり(右)、CsFLの機能が喪失していると考えられました。
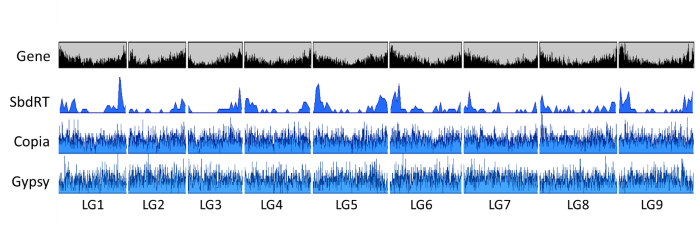
図5 全染色体におけるレトロトランスポゾンと遺伝子の分布。CopiaタイプのレトロトランスポゾンとGypsyタイプのレトロトランスポゾンは9本の染色体(LG1~LG9)にあまり偏りなく存在している。遺伝子は染色体の端部領域に多い傾向がみられるが、SbdRTの分布はこれに非常によく似ている。縦軸は‘密度’、横軸はそれぞれの染色体上の位置を示す。
【今後の展開】
・キクタニギクのゲノム情報を元に栽培ギクにおける農業上重要な遺伝子が明らかにされることが期待されます。また、ゲノム情報を用いた選抜手法が栽培ギク育種にも適用することができるようになります。
・キクタニギクを用いて明らかにされる重要遺伝子をゲノム編集などにより改変した全く新しい栽培ギクが育成されることが期待されます。
・頭状花序などのキク科に特徴的な性質を制御する遺伝子が明らかになることが期待されます。
(*1)六倍体:多くの生物は二倍体であり、ゲノムのセットを二つ持つが、六倍体は6セット持っており、ゲノムの構成は複雑になる。
(*2)キク連:栽培ギク・キクタニギクが属すキク属を含む、より広範な分類群。ヨモギ・除虫菊・春菊などを含む。
(*3)レトロトランスポゾン:いわゆる動く遺伝子(トランスポゾン)の一種。レトロトランスポゾンは染色体上を動く際に、元のコピーも維持されるため、転移するたびにコピー数が増える。そのため、大きなゲノムサイズの生物には多数コピー存在することが多い。
(*4)自家不和合性:自身の花粉がめしべについても受精が起きず、種子が形成されないようにする仕組み。
(*5)ヘテロ性:ひとつの個体内の同じ遺伝子に異なる型(塩基配列)のものを持つ傾向。二倍体なら同じ遺伝子を二つ持つが、一般的には塩基配列は同一とは限らない。一方、純系では全ての遺伝子は同一の塩基配列を持つ。
(*6)HiSeq:ショートリードシーケンス(短い塩基配列)を大量に解読する次世代シーケンサーの一種。
(*7)PacBio sequel:ロングリードシーケンス(長い塩基配列)の解読が出来る次世代シーケンサーの一種。
(*8)Hi-C法:染色体の高次構造の解析法。塩基配列断片の整列化にも有用な解析法。
(*9)ドラフトシーケンス:塩基配列としては全ゲノムの大部分をカバーしているが、短い断片配列のままで、染色体レベルでひとつながりにつながっていない。
(*10)CsFL:花発生のマスター制御因子LEAFYのキクタニギクにおけるカウンターパート。
(*11)頭状花序:小さな花が集まって作られる、ひとつの大きな花のような構造。キク科の特徴的な花序形態。
本研究はNBRPゲノム情報等整備プログラム「ロングリードを用いたキク属モデル系統のゲノム解析」による研究助成を受けて行われました。
<研究に関すること>
広島大学大学院統合生命科学研究科
教授 草場 信
Tel:082-424-7490 FAX:082-424-0738
E-mail:akusaba*hiroshima-u.ac.jp
<広報に関すること>
広島大学財務・総務室広報部広報グループ
Tel:082-424-3749 FAX:082-424-6040
E-mail:koho*office.hiroshima-u.ac.jp
(注: *は半角@に置き換えてください)
掲載日 : 2021年10月27日
Copyright © 2003- 広島大学