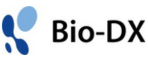広島大学大学院統合生命科学研究科 教授 坊農秀雅
Tel:082-424-4013
E-mail:bonohu*hiroshima-u.ac.jp
(*は半角@に置き換えてください)
本研究成果のポイント
- サンガーシーケンス(*1)データからゲノム編集の結果生じた挿入・欠失変異(インデル)(*2)を広範囲(最大約200塩基対)にわたり推定可能な解析ソフトウェア「PtWAVE」を新たに開発しました。
- PtWAVEは、既存手法では検出が困難だった50塩基対を超える大きな欠失変異を含むDNAサンプルにおいて、従来のTIDE解析(*3)を実施するソフトウェアと比べて優れた精度と感度を示しました。
- 本ソフトウェアは商用利用を含め誰でも無償で利用可能であり、ウェブ上で公開されています。これにより、大規模インデルを含むゲノム編集(*4)の結果を高精度に評価できるようになり、ゲノム編集研究の効率化や安全性評価の向上に貢献することが期待されます。
概要
広島大学ゲノム編集イノベーションセンタープラチナバイオ共同研究講座の中前和恭共同研究講座助教、広島大学発スタートアップ企業であるプラチナバイオ株式会社の奥原啓輔CEO、中川佳子主任研究員、伊出佐耶研究員、大貫永輝研究員、そして坊農秀雅教授(広島大学大学院統合生命科学研究科・プラチナバイオ共同研究講座教授兼任)らは、ゲノム編集により生じる大規模な挿入・欠失変異(インデル)をサンガーシーケンスデータから高感度に推定できる新しい解析ソフトウェア「PtWAVE」(Progressive-type Wide-range Analysis of Varied Editsの略)を開発しました。本研究により、これまで検出が難しかった50塩基対を超える大きな欠失変異を含むゲノム編集の結果を正確に解析する手法が提供され、ゲノム編集技術の評価・応用に新たな展開をもたらすことが期待されます。
本研究成果は、学術雑誌「BMC Bioinformatics」に4月29日付けで掲載されました。
発表論文
- 論文タイトル:PtWAVE: a high-sensitive deconvolution software of sequencing trace for the detection of large indels in genome editing
- 掲載雑誌:BMC Bioinformatics
- DOI番号:https://doi.org/10.1186/s12859-025-06139-8
- 著者:Kazuki Nakamae※1,2, Saya Ide1,2, Nagaki Ohnuki1,2, Yoshiko Nakagawa2, Keisuke Okuhara2,3, Hidemasa Bono※1,3
※責任著者
|
1 広島大学ゲノム編集イノベーションセンター |
| 2 プラチナバイオ株式会社 | |
| 3 広島大学大学院統合生命科学研究科 |
背景
TIDE解析は、サンガー法によるDNAシーケンス波形データを計算的に分解してゲノム編集ツールによる標的変異導入効率を推定する手法であり、ゲノム編集分野で重要な役割を果たしてきました。しかし、従来のTIDE解析では解析可能なインデルの大きさに制限があり、既存のTIDE解析ツールのほとんどはより大きな欠失や挿入変異(50塩基対以上)の検出には対応できませんでした。解析範囲を拡大すれば理論上は大きな変異を検出できますが、その場合は考慮すべき変異パターンの増大により解析結果の不確実性が高まることが課題でした。また、50塩基対を超える大きな欠失変異に対する既存のTIDE解析ツールの精度や感度についても十分に検証されていませんでした。このため、ゲノム編集によって生じる大規模インデルを正確に推定し、定量を行うことができる新たな解析手法の開発が求められていました。
研究成果の内容
本研究では最大約200塩基対のインデルまでの幅広いサイズのインデルを解析可能な新規ソフトウェア「PtWAVE」を開発しました(図1)。解析アルゴリズム上の特徴として、モデルの不確実性を抑えるために可変選択のモードやフィッティングアルゴリズムを選択できるようにしており、従来手法では解析精度が低下する可能性があった部分に対策を施しています。これにより、インデル混合状態のシーケンス波形データの分解を安定して行い、編集サンプル中の各種インデルの割合や編集効率を算出することが可能になりました。
広島大学では開発したPtWAVEの性能を評価するため、85 塩基対の欠失変異を含む人工合成したDNAサンプルを用いた検証を行いました。その結果、PtWAVEは85 塩基対の欠失変異の割合を高い精度で再現良く検出できることを確認しました。一方、従来のTIDE系ツールであるICEやTIDEでは、85塩基対の欠失変異をほとんど検出できないことが改めて示されました。
また、50塩基対以上の欠失についても対応可能な既存ツールであるDECODRについてもPtWAVEとの性能比較を行ったところ、DECODRは一部の条件では85塩基対の欠失に優れるものの、総合的にはPtWAVEが最も優れた成績を出力できることを示しました。85塩基対の欠失変異の検出精度(Accuracy)に関してはDECODRの各解析モードにおいても解析精度が高い(R2値が0.8以上)とされたサンプルでも88%程度なのに対し、PtWAVEの「all」解析モードでは100%を記録し、12%の精度向上を確認しました(図2)。
このことから、PtWAVEは既存の解析アルゴリズム上の課題を克服し、大きな欠失変異が含まれるデータに対しても頑健な解析が可能であるといえます。 さらに、実際のゲノム編集サンプル由来のサンガーシーケンスデータについてPtWAVEを適用したところ、人工合成DNAサンプルでの検証と同様に安定した解析結果が得られることを確認しました。以上の検証から、PtWAVEはインデル混在サンプルの解析において広範囲の変異を高感度に検出でき、特に大規模欠失変異の検出能力に優れていることが示されました。
PtWAVEはウェブブラウザ上で動作するオンラインツールとして、広島大学発スタートアップ企業であるプラチナバイオ株式会社より商用利用問わず無償で提供されています。
- PtWAVE: https://www.ptwave-ptbio.com
今後の展開
本研究で開発されたPtWAVEは、ゲノム編集における大きな欠失変異を伴う解析を可能にする点で画期的であり、今後の様々な応用が見込まれます。特に、ゲノム編集で大規模な欠失変異が起こりやすい生物種や細胞系統において、本ツールが解析の標準手段として活用されることで研究の効率化が期待されます。また、従来の手法では見逃されていた大規模なオフターゲット変異や予期せぬゲノム改変を推定できる可能性が高まるため、遺伝子治療などゲノム医療の安全性評価にも寄与すると考えられます。今後は、実験系の種類や生物種を問わず幅広い場面でPtWAVEの有用性を検証され、解析アルゴリズムの更なる改良を進めながら、ゲノム編集研究の加速と安全な応用開発に貢献していくことが期待されます。
参考資料
図1
PtWAVEのユーザインタフェース。(A)入力パラメータ設定画面です。編集標的周辺のシーケンストレースとプロトスペーサー配列、PAM配列、検出するインデル範囲を指定することができます。(B)検出されたインデルサイズの分布を示す棒グラフです。(C)編集効率(editing_eff)と解析評価指標(BIC、r_sq)の表示です。(D)予測された各アレルの貢献度(頻度)を示す結果画面です。
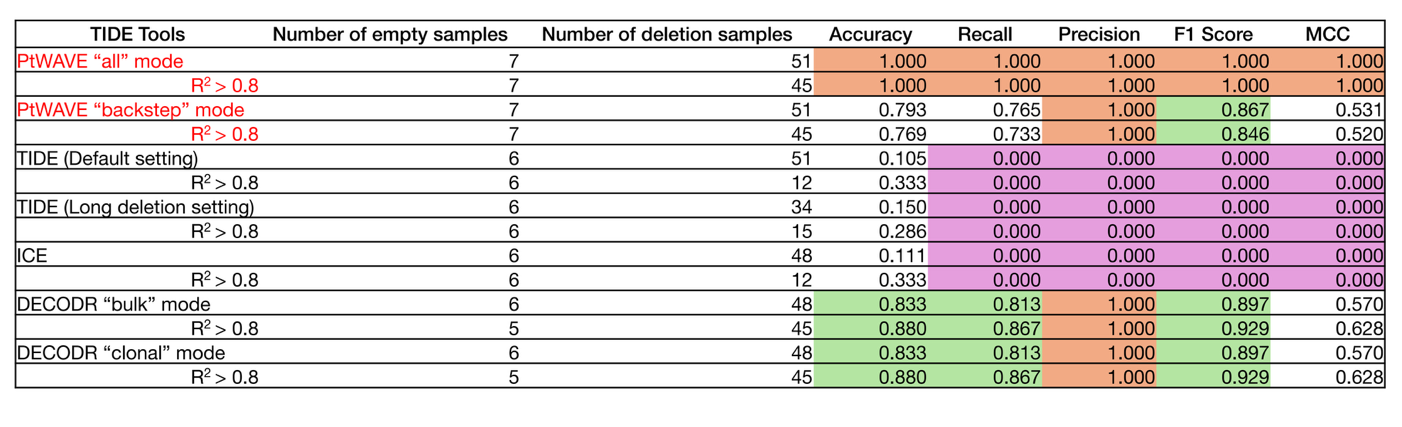
図2
各種TIDE解析ツールによる大欠失変異検出性能の比較。85 bp欠失の有無を検出するタスクについて、各ツールの精度指標を比較した表です。解析が正常に完了したempty(陰性)またはdeletion(陽性)サンプルの数とともに、Accuracy(正解率)、Recall(再現率)、Precision(適合率)、F1スコアといった各種精度指標の値を記載しています。赤字で示されたものがPtWAVEの結果であり、all解析モードにおいては特に正解率、再現率、適合率、F1スコア、MCCの全てが1.000(100%)と最高性能を記録している(表中オレンジ色欄)。対照的に、TIDEやICEでは欠失検出ができないためにAccuracy以外の精度指標が0となっている(表中紫色欄)。DECODRではMCC以外の指標で0.8以上(表中緑色欄またはオレンジ色欄)を示すものの、PtWAVEの最高値(「all)解析モードの結果)と比べると総合的に低いことが確認できます。
用語説明
*1 サンガーシーケンス(サンガー法): DNAの塩基配列決定法の一つ。蛍光標識した核酸塩基を用いてDNA合成反応を停止させ、そこから形成される塩基ごとの波形データから配列を読み取る手法です。現在でも小規模なDNA配列の解読やゲノム編集の活性確認に広く利用されています。
*2 挿入・欠失変異(インデル):核酸塩基のInsertion(挿入)とDeletion(欠失)による塩基配列変異の総称。一つ以上の塩基がゲノムDNA上で新たに挿入されたり欠落したりする変異を指します。塩基の欠失が起こる変異は特に「欠失変異(deletion)」とも呼ばれることもあります。
*3 TIDE解析:Tracking of Insertions and DEletions解析の略称。ゲノム編集ツールによって導入された標的変異の効率を推定するために、サンガーシーケンスの波形データを解析する手法、あるいはそれを実行するプログラムを指します。従来から広く利用されていましたが、解析可能なインデルサイズに制限があることが知られています。代表的な関連ツールとして、同様の目的で開発されたICE(Inference of CRISPR Edits)やDECODR(Deconvolution of Complex DNA Repair)などがあります。
*4 ゲノム編集:細胞内にある生物の遺伝情報(ゲノム)上に存在するDNA配列に対してヌクレアーゼ(DNA 切断酵素)等を部位特異的に作用させることで遺伝子を改変する技術を指します。
~研究のポイントをやさしく解説!~
広島大学とプラチナバイオ株式会社の研究チームが、ゲノム編集によってDNAがどのように変化したのかを、より正確に調べることができる新しいソフト「PtWAVE(ピーティーウェーブ)」を開発しました。
このソフトは、特にDNAの50文字以上の大きな変化(欠失)も見つけられるのが特徴で、これまでの方法では検出が難しかった部分までしっかり調べられます。
1. 研究の背景と目的
- ゲノム編集は、DNAの一部を書き換える技術で、医療や農業など様々な分野で注目されています。
- 編集後に生じる予期しない変化(特に大きなDNAの欠失や挿入)が正確に検出できていないという課題がありました。
- 今回の研究では、これまで検出が難しかった大きな変化を高精度に解析するための新しいソフトを開発しました。
2. 開発したソフトウェア「PtWAVE」とは?
- 名称:「PtWAVE(ピーティーウェーブ)」
- 正式名:Progressive-type Wide-range Analysis of Varied Edits(さまざまな編集変異を広範囲・段階的に解析する手法)
- 主な機能:
o 従来の解析手法では見つけづらかったDNAの50塩基対以上の大きな変異を検出
o 古くから使われているサンガーシーケンスのデータに対応しており、手軽に利用可能
3. 従来技術との違い
- 一般的な解析ソフトでは、小さな変異(数塩基の欠失や挿入)にしか対応できないことが多かった。
- 「PtWAVE」は、大きなインデル変異(挿入・欠失)の検出に強く、より正確なゲノム編集の評価が可能。
4. 研究体制と関係者
- 開発チーム:
o 中前 和恭 共同研究講座助教(広島大学ゲノム編集イノベーションセンター/プラチナバイオ共同研究講座)
o 奥原 啓輔 CEO、中川 佳子 主任研究員、伊出 佐耶 研究員、大貫 永輝 研究員(プラチナバイオ株式会社)
o 坊農 秀雅 教授(広島大学大学院統合生命科学研究科・プラチナバイオ共同研究講座)
5. 社会的意義・今後の展望
- 「PtWAVE」は、ゲノム編集の活性検証や安全性評価において、新しい標準ツールになる可能性がある。
- 特に、医療応用における正確な変異解析や、農作物の品種改良などへの貢献が期待される。

 Home
Home